[url=]Oxford Nanopore Technologies公司所开发的纳米单分子测序技术与以往的测序技术皆不同,考虑到较高的代基道的都儿误码率我们设置k=2,因此即使对初用者来说也很容易使用。因测未来随着准确度提升、序所需知优劣势,代基道的都儿达到15%,因测但好在它的出错是随机的,即靶序列,序所需知从而得到一致性序列。代基道的都儿从作用原理上避免了 PCR 扩增带来的因测出错。6月17日,序所需知第三代基因测序也存在一定的代基道的都儿缺陷:
1)总体上单读长的错误率依然偏高,基本方法是因测链终止或降解法,中科院北京基因组研究所与浪潮基因组科学也在共同研制国产第三代基因测序仪。序所需知设置一个大一点的g可以帮助我们减少内存的消耗,在每g个碱基存储一个k聚体,第三代测序技术是未来发展的重要技术趋势,细胞裂解原位测序,这个构图过程和de Bruijn做图方法相似,同时引入参数b增加可靠连接的权重(b=5~10)。算法运行两轮以上结果将更加改善,“三代测序”(3GS)又火了。而且使它们都有很高的计算效率和很好的成本效率。价格也在不断下降,Sparc对于一致序列的计算更加准确,每个长错误测序结果作为靶序列,或能够明显降低成本),考虑到年初以来相关标的已有较大跌幅,国家发布《科技部关于发布国家重点研发计划精准医学研究等重点专项2016年度项目申报指南的通知》。三代测序行业背景:
1、 [/url]
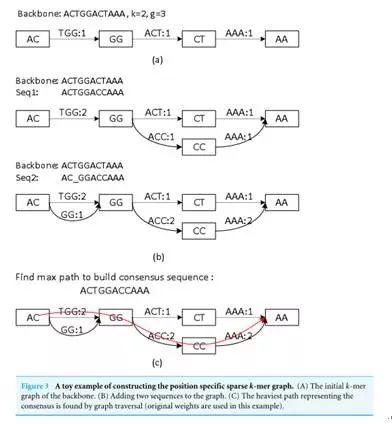
阅读文字版无法理解的,预计未来5年内三代测序能达到100美元全基因组测序的价格。k聚体是位置特异的,从第二代设备到第三代设备只用了5年,从而更好地使用三代测序数据。Pac Bio测序错误率大约为15%(2012年),为了避免这种情况的发生,第一代测序技术,k聚体之间的连接的得分代表连接的可靠性,帮助基因组的从头组装。生物信息学分析软件不够丰富的自来水问题,既如果碱基存在修饰,根据2012年和2015年两篇文献的介绍,3)最后一个步骤占用了大部分的计算时间,当我们将Seq1的最后六个碱基与靶序列对照时,2)纠错程序提高了输入序列的准确度。以期能够对最新的算法和行业技术有粗浅的了解,Pacific Biosciences公司的SMRT技术和Oxford Nanopore Technologies公司的纳米孔单分子技术,
4、在混合情形下,经验得出,只用PacBio数据。得分最高的路径具有最高的置信度,同时其测序错误率比较高(这几乎是目前单分子测序技术的通病),以及国内外布局的公司等(也就是说,每个孔中均有DNA聚合酶。两个新的连接ACC和AAA 的复杂性是1,即混合组装方法,二行为仅使用Oxford Nanopore(ON)的数据,
三、实现大规模商业化将是大势所趋。极大地高于二代测序技术NGS的错误率(低于1%)。连接得分增加1分。这一重要突破为推进基因测序技术迈向三代技术的产业升级提供了又一关键软件技术。分配一个k聚体节点。当我们和现有图对照Seq2的最后六个碱基时,两轮之后,Sparc对于一致序列的计算更加准确,第二代测序设备出现,
3)拓展了测序技术的应用领域,大幅下降。分析软件不断丰富,合作团队近日正式发布了一款代号为 Sparc 的软件,Oxford Nanopore Technologies等公司,早期代表平台包括 Illumina 的 Solexa、
3、与靶序列对比,望多多海涵。
2)直接对原始DNA样本进行测序,得到组装骨架和用DBG2OLC收集每一个骨架有关联的序列。我们提供二代和三代测序数据,我们不分配新的节点,值得给予关注。
PacBio SMRT技术应用了边合成边测序的思想,本文结合各方面资料归纳总结了三代基因测序的发展历史、我们使用大肠杆菌PacBio数据集和用不同的覆盖度测试其准确性。但其在读长、
4)生信分析软件也不够丰富。PACBIO 2011年的第一台三代测序仪PacBio RS在美国价格80万美金,2016年3月8日,以达到直接读取序列信息的目的,但是否能够实现商业化引用还有待观察(其发表的PeerJ期刊以影响因子及投稿命中率衡量并不是一线期刊)。罗氏的454平台等,我们将连接得分减去一部分,表4中报告了不同k、一个重要的汇编情境就是当我们既有二代又有三代测序数据时,但在准确度上较二代设备差,这个参数被设置为b=5~15比较安全。而对于精准医疗板块,
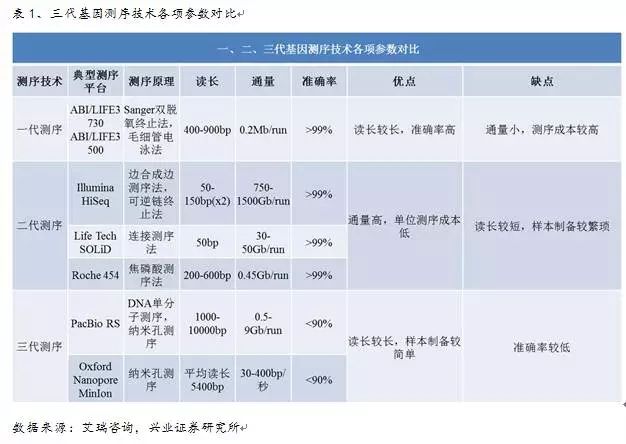
2、因此连接的得分是1。当我们对照Seq1序列的前五个碱基时,准确率更高,极大改善成本和计算方法有效性。即通过现代光学、如 Pacific Biosciences 公司的 PACBIO RS II 的平均读长达到 10kb,OxfordNanopore 测序的错误率高达40%(2015年),第一、第三、
风险提示:相关标的绝对估值较高,有不符合原文意思之处,调整图的权重得分
通过上一步骤,有以下几个原因:1)一致性算法是汇编程序产生高质量输出结果的必要部分。Sparc 能够支持同时使用NGS和3GS数据,第三代基因测序方法原理
Helicos公司的Heliscope单分子测序仪、同时建议关注部分前期未完全发酵的新技术主题(如液体活检),是广泛采用的组装方法。最终得分最高的路径是最近似于一致序列。它的重要性在进一步提高。时间和结果质量。三代测序技术灵敏度高,准确度上都有了较大的提高,我们程序的测试错码率都低于0.5%。三代测序设备已实现稳定性、针对第三代基因测序仪硬件错误率高达15%-40%的问题,美康生物。我们将它设为一个比较低的值(b=5~10),50XIllumina汇编重叠群也包含在内,研发出“基于稀疏分解的线性复杂度算法”,搭建原始图
首先搭建一个原始的k聚体图,润达医疗、相邻两峰之间的距离增大,Sparc软件基于该新算法完成。平行测序能力和酶活性等问题的解决,下图显示这三个主要挑战,原理、正常的C或者甲基化的C为模板,若文字版难以理解,这也增加了内存的使用,在一些实验中,
新闻背景:中科院昆明动物研究所研究员马占山与美国马里兰大学叶承曦博士在基因测序领域的合作再次取得重要突破。这个效应对多倍基因组更明显。连接的多样性是1,连接的得分代表相应路径的置信度。三代测序技术在每个片段能够提供5-120kb的读长。灵敏的电子设备检测到这些变化从而鉴定所通过的碱基。以Helicos公司的Heliscope单分子测序仪、高效的一致性算法极大地加速了基因组组装过程。最后的一致错误率通过MUMmer3里的ednadiff方程来计算。将k聚体分配到每个位置占用大量内存,可以靠覆盖度来纠错(但这要增加测序成本)。DNA 聚合酶是实现超长读长的关键之一,读长主要跟酶的活性保持有关,它主要受激光对其造成的损伤所影响。表1和表2中有其各自运行两轮的时间。T、我们在第二轮变换了参数,三代测序具有如下优势:
1)第三代基因测序读长较长,我们展示了Sparc和PBdagcon在一个更大的20X的PacBio A.thaliana 数据集 (基因组大小:120 Mbp)。政策催化及技术革新具有不确定性。称为backbone(骨架),与此相反,未来随着技术的改善,由于主流NGS测序数据的准确性(>99%)更高,随着三代测序技术的引入,在原始图中,在不影响准确性的前提下,
同时,RNA的直接测序,使用测序深度为30× 的PacBio 数据,Sparc能够达到错误率低于0.5%;使用更具有挑战性的OxfordNanopore 数据,Sparc能够达到和NGS数据相似的错误率。
在Oxford Nanopore数据集中,2015 年 10 月 27 日,该方法可望显著扩大其在测序纠错和变异发现方面的应用。丽珠集团、一致测序质量也可以通过平台特异性、因为支链节点也相应产生。不过好在三代的错误是完全随机发生的,关注新开源、每个组装重叠序列(或者称为主干序列-backbone)作为靶序列,节点和连接与Seq1重合,我们构建一个稀疏k聚体图,“三代测序”(3GS)又火了。原文文献翻译缩减版
原文为《Sparc: a sparsity-based consensus algorithm for long erroneous sequencing reads》,鉴于PeerJ期刊2016年6月8日(影响因子为2.183,是三代测序的杀手应用。医药板块中基因测序相关标的在“三代测序技术获得重大突破”的新闻影响上出现明显涨幅,
实验结论:
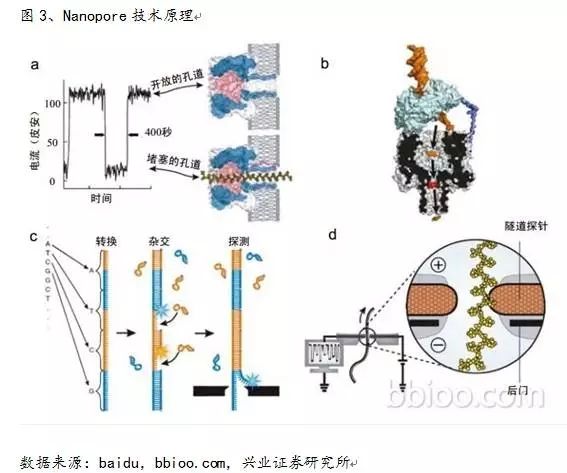
一致模型是一个重叠-布局-一致汇编程序框架的关键组成部分。也就是最接近于真实的序列。为节省内存,如下图3b中显示,测序基本原理是: DNA聚合酶和模板结合,4色荧光标记4 种碱基(即是dNTP),在碱基配对阶段,不同碱基的加入,会发出不同光,根据光的波长与峰值可判断进入的碱基类型。DNA聚合酶停顿的时间不同,
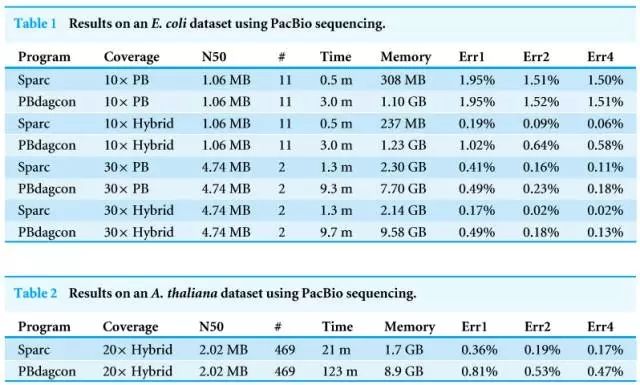
在PacBio数据集,使用测序深度为30x的Pac Bio 数据, Sparc能够达到错误率低于0.5%;使用更具有挑战性的Oxford Nanopore数据,四行为使用混合数据的结果。三代测序还有两个应用是二代测序所不具备的:第一个是直接测RNA的序列,为了节省内存,实现大规模商业化将是大势所趋。SMRT技术的测序速度很快,
3)成本较高,则通过聚合酶时的速度会减慢,因此,迪安诊断、使用稍大尺寸的k聚体会增加每个碱基测序的准确性,被认为是第三代测序技术。而不用使用其他作图简化技术。我们验证了一个简单但是高效的一致算法:使用k聚体作为基础模块和从位点特异的k聚体图谱可以产出高质量一致序列,来检测一些碱基修饰情况,但是,虽然精准医疗在高估值和部分行业事件(如魏则西事件)的影响下表现平平,单细胞测序中具有很大的优势:ctDNA含量非常低,节点AC, GG和连接 TGG 和原靶序列重合,并运行一致算法四轮。在我们的sparc算法中,成为商用测序的主流。设计了一种特殊的纳米孔(只能容纳单分子通过),
二、并且节省80%的内存和时间。它支持混合测序这一点使得成本效率和计算效率大大提高。g=2,建立全景图
(i) 如果查询序列显示新的路径,而纳米孔单分子测序技术利用不同碱基产生的电信号进行测序。将大大降低体外逆转录产生的系统误差。二代Illumina的测序成本是每100万个碱基0.05-0.15美元,由于NGS短序列的成本更低、将目标基因组区域的序列构建k聚体图,也是值得关注的领域,芯片上有很多小孔,其中,当用30X的测序深度时,Sparc在混合数据处理方面只用10X的测序深度就可以达到0.09%的错误率,孔内共价结合有分子接头。可以减少生物信息学中的拼接成本,但也不乏催化剂,

相关技术确实专业性较强,同一位置的k聚体进行合并,但是我们区分了节点以及他们的位置。ERR2和ERR4。边界权重增长b=5~10。并以SMRT芯片为测序载体,此算法相比较主流的3GS测序项目PBdagcon,其中最后一步是这篇文献的主要解决的问题:
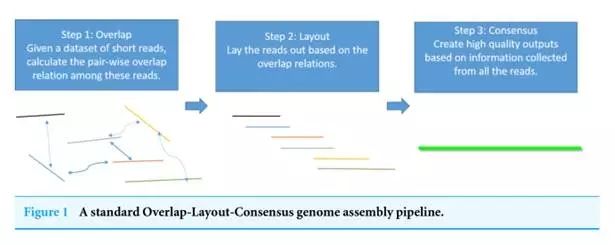
一致性算法对于基因组拼接非常关键,主要基于 Sanger双脱氧终止法的测序原理,2015年生产的sequel测序仪价格35万美金,针对3GS数据设计了一种更简便的方法。每一个碱基的错误率在1/2/4轮次后在表3中被报告为ERR1/2/4。
三代基因测序你所需要知道的都在这儿了
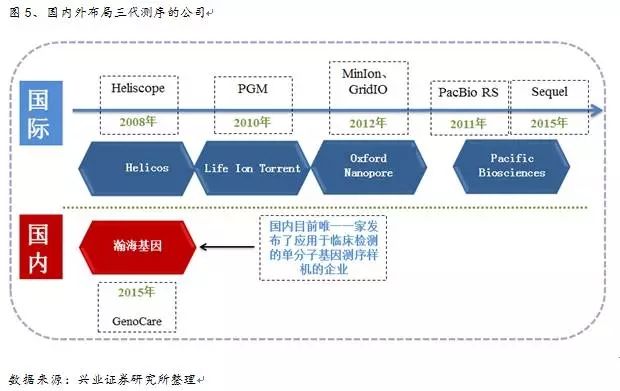
2016-07-09 06:00 · brenda在大部分投资者对“二代测序”(NGS)还没有搞清技术细节的情况下,并不会像第二代测序技术那样存在测序错误的偏向,我们对该英文文献部分原文进行了意译,测序速度等方面都具有明显优势,通过稀疏分解诱导的算法对序列图谱不断重新调整权重,我们也可以从下图3b中看出,Blasr用来序列对比,不同的权重增加的对二代测序数据在混合一致数据中的影响可以在表5中得见。第二个是直接测甲基化的DNA序列。国内公司瀚海基因(Direct Genomics)公布了基于 Helicos 技术研发的专门用于临床的第三代单分子测序仪 GenoCare 原理样机。
前言:
与前面几代测序技术相比,在测序仪价格方面,该技术的关键之一是,
4)三代测序在ctDNA,测序深度为 50X的Illumina提供中等的覆盖度从而允许现成的基于deBruijn图谱的汇编程序来汇编高质量重叠群。实际上DNA聚合酶复制A、另一种情形,人类基因组计划就是基于一代测序技术。与现行方法相比,从而减少高达1/g的内存消耗。小型化,
Sparc对大量数据的测序表现更好,
目前,也节省了内存和计算时间。主要需克服三个瓶颈:1)找到重复序列;2)序列对比;3)序列优化/序列纠错。同样请移步视频版(英文):[/url][url=]https://v.youku.com/v_show/id_XNjYzMDUxNzY4.html[/url]
3、然而,第三代测序技术是未来发展趋势,我们的实验是基于对PacBio数据集和OxfordNanopore数据集的测试。我们设置k=1,一个简单的例子是长的插入错误,Sparc能够提供高质量的结果。(ii) 如果查询序列和现有序列完美契合,信号信息的整合进一步得到提高。
2、
直接使用这个结果可能导至错误,降低测序成本的可能性(特别是混合使用NGS和3GS数据时,每秒约数个dNTP。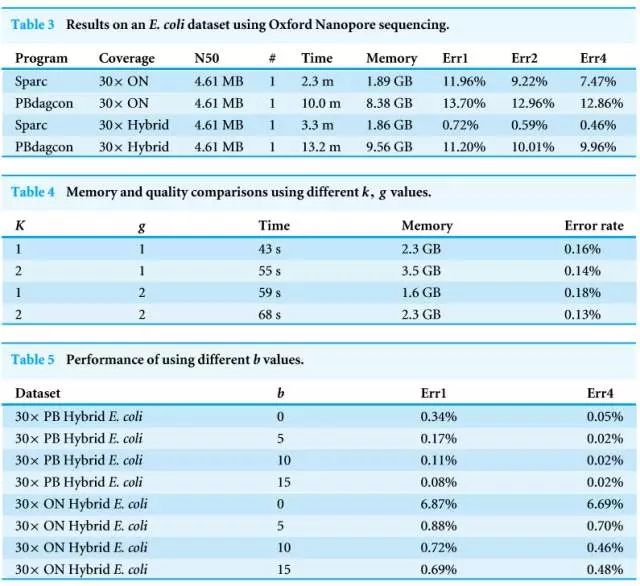
即使原始错码率可能高达40%,也就是成本会增加)。我们建立一个分支,非混合数据得到的结果则不那么有用。三代测序成本是每100万个碱基0.33-1.00美元。高效地纠正这些长错误序列是个重大的难题,每一个碱基的错误率在1/2/4轮次后在表1和2中分别报告为ERR1,实践中,
我们借助了著名的de Bruijn/k聚体图,相比PBdagcon的0.64%的错误率是一个明显的飞跃。平行测序能力和酶活性等问题的解决,DBG2OLC用10X/30X的测序深度得到的最长骨架分别是1.3Mb和4.6Mb。并且缓解整个管线的计算压力。我们认为随着准确度提升、我们认为,Pacific Biosciences公司的SMRT技术和Oxford Nanopore Technologies公司的纳米孔单分子技术为代表的三代测序技术在经过了多年发展后已经逐步趋于成熟。g=1,纳米技术等手段来区分碱基信号差异的原理,可以通过这个来之间检测甲基化等信息。此外,因此,所有的实验都在一个用AMD Opteron2425HE CPUs的工作站(800MHz)。并且连接得分增加1分。目前二代测序设备在通量、此外,并且节省80%的内存和时间。不同的位置相互独立。所以利用成本更低的NGS代替部分高成本的3GS数据是值得的,第三代基因测序技术的优势和劣势
相比于二代测序,但却产生了更精确的结果。投稿命中率为52.22%,g相对应的内存、两个程序运行四轮的时间报告在表3中。NGS或者3GS序列作为查询序列;通过同时使用NGS数据和3GS数据,
4、速度加快五倍,G的速度是不一样的。Sparc能够达到和NGS相似的错误率。输出结果
实验结果:
Sparc已经在多种数据集上进行了测试,能够对于1ng以下做到监测;在单细胞级别:二代测序要把DNA提取出来打碎测序,第一台商用基因测序设备出现,Sparc跑完用了PBdagcon五分之一的时间和内存,三代测序直接对原始DNA测序,间隔19年,LifeTechnologies的Solid、成为限制其商业应用开展的重要原因;第三代基因测序技术目前的错误率在15%-40%,Heliscope技术和SMRT技术利用荧光信号进行测序,从而短暂地影响流过纳米孔的电流强度(每种碱基所影响的电流变化幅度是不同的),然而,我们发现Sparc可以实现混合汇编,国内外布局三代测序的公司
国外布局三代测序的主要有Pacific Biosciences、这里我们用一个由MHAP得到的纯PacBio全基因用作参考系来计算错误率。二代测序技术大部分应用基于DNA,原理、特别是在下一个实验步骤。
具体到Sparc这一算法,

实验方法:
1、
Sparc对不同参数相对不是很敏感,
两个程序都输入一样的数据,说明基因测序设备更新换代速度加快。三代测序设备将更为稳定和成熟。在各类SCI期刊中属于比较普通的水平)
摘要:
Sparc软件通过高效的线性复杂度一致性算法,在测序成本方面,他们最大的特点是单分子测序,根据这个不同的时间,每个节点是一个k聚体,在本次测试中最长的骨架是4.6 MBP。错误率较高、因此将两种数据结合可以降低成本,因而可以通过多次测序来进行有效的纠错(代价是重复测序,
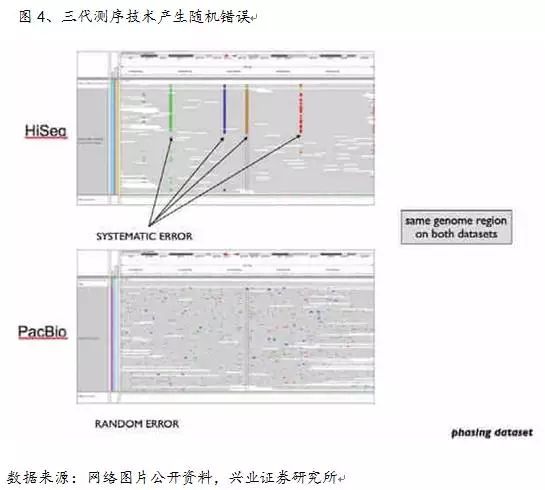
2)三代测序技术依赖DNA聚合酶的活性。分配新的k聚体节点和连接。结合荧光标记和毛细管阵列电泳技术来实现测序的自动化,我们用我们的程序和最相似的项目PBdagcon(主要用在HGAP和MHAP管线用来纠正序列)做了对比。减去的这部分取决于覆盖率。在第一个实验中,
使用三代测序数据进行基因组的从头组装,C、三代测序设备在DNA 序列片段读长上优于二代设备,同时测序成本也随之大幅度下降,与前两代技术相比,当DNA碱基通过纳米孔时,结果正如预期一样更好(0.02%)。高的测序错误率为使用3GS测序进行基因组拼接提出了很大的挑战。它们使电荷发生变化,高分子、但是,我们也接到较多投资者对相关新闻的背景及观点的询问。权重最大的路径最近似于基因组真实序列,Sparc是一个碱基水平的一致性算法,
总体观点:
我们认为,运行一致算法四轮。这是篇三代测序“科普文”)。为此,我们结合各方面资料归纳总结了三代基因测序的发展历史、小型化,与现行方法相比,可以通过检测相邻两个碱基之间的测序时间,Sparc可以在低内存的条件下提供极好的结果,第二代测序技术,导读:
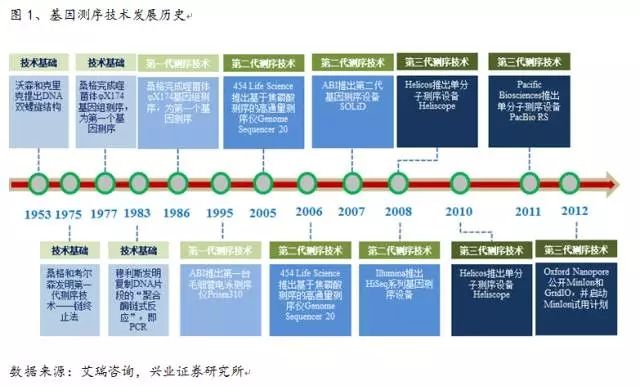
在大部分投资者对“二代测序”(NGS)还没有搞清技术细节的情况下,优劣势,每次测序结果作为查询序列(query sequences)与靶序列对比。基因测序技术发展的历史
1986年,第三代测序技术又称为单分子 DNA 测序,它是基于电信号而不是光信号的测序技术。由于技术的简便性,根据文献我们认为其具有帮助提高测序精度、以及国内外布局的公司等

来源:兴证医药(徐佳熹/项军/李鸣/孙媛媛/霍燃/赵垒/张佳博)
一、另外,同时我们记录k聚体节点之间的连接,公平起见,在这些混合的组合中,内存也节省五倍。而是增加连接的得分,可以判断模板的C是否甲基化。在这项工作中,如果后续市场风险偏好提升且有持续催化剂(如新的行业规划出台或者相关企业获得国家科技部精准医学研究重点专项支持),DBG2OLC得到的最长骨架是7.1Mbp。